Перейти к:
Хемотранскриптомный анализ эффектов молекулы 7-гидроксиматаирезинола на опухолевые клетки молочной железы человека линии MCF7
https://doi.org/10.17749/2313-7347/ob.gyn.rep.2023.409
Аннотация
Введение. Установление эффектов воздействия онкопротекторных средств на транскриптом – важная процедура фармакологии, необходимая для комплексной оценки желательных и нежелательных свойств препаратов-кандидатов.
Цель: оценка транскриптомных эффектов лигнана 7-гидроксиматаирезинола (7ГМР) по отношению к клеткам опухоли молочной железы.
Материалы и методы. Хемотранскриптомный анализ 7ГМР проведен для клеток линии MCF7 (опухолевые клетки рака молочной железы) в условиях инкубации с клетками в течение 24 ч. В базе данных GEO (англ. Gene Expression Omnibus) находятся выборки данных транскриптомных исследований, и на основании алгоритмов комбинаторного анализа хемографов моделируется дозозависимое воздействие молекулы на экспрессию генов. В результате формируются списки генов с изменённой экспрессией, которые анализируются методом функционального связывания с использованием международной номенклатуры Gene Ontology (GO) по биологическим ролям генов/белков.
Результаты. Установлены дозозависимые эффекты воздействия 7ГМР на транскрипцию генов (изменение транскрипции на 5 % или более на 1 мкмоль 7ГМР) для 3468 из 12700 исследованных генов: 7ГМР достоверно снижал экспрессию генов, вовлечённых в поддержание пролиферации клеток (401 гена, в том числе генов, участвующих в поддержании теломер), синтез белка (194 генов) и протеасомную деградацию белков (70 генов), энергетический метаболизм опухолевых клеток (91 гена) и хроническое воспаление (148 генов). Уменьшение экспрессии этих групп генов тормозит процессы пролиферации и жизнедеятельности, одновременно защищая организм от избыточного воспаления. 7ГМР способствовал преимущественному повышению транскрипции групп генов, вовлеченных в онкопротекторное действие (более 100 генов), в том числе генов, участвующих в поддержании онкопротекторного иммунитета, а также генов, опосредующих онкопротекторные свойства витамина D, ретиноидов и витамина С.
Заключение. Выявленные изменения в транскрипции генов усиливают воздействие 7ГМР на белки протеома и указывают на перспективы использования 7ГМР для эффективной и безопасной профилактики и терапии узловой мастопатии и рака молочной железы.
Ключевые слова
Для цитирования:
Торшин И.Ю., Рубашкина А.Н., Громова О.А. Хемотранскриптомный анализ эффектов молекулы 7-гидроксиматаирезинола на опухолевые клетки молочной железы человека линии MCF7. Акушерство, Гинекология и Репродукция. 2023;17(5):584-596. https://doi.org/10.17749/2313-7347/ob.gyn.rep.2023.409
For citation:
Torshin I.Yu., Rubashkina A.N., Gromova O.A. Chemotranscriptomic analysis of 7-hydroxymatairesinol-related effects on MCF7 human breast tumor cells. Obstetrics, Gynecology and Reproduction. 2023;17(5):584-596. (In Russ.) https://doi.org/10.17749/2313-7347/ob.gyn.rep.2023.409
Введение / Introduction
Лигнан 7-гидроксиматаирезинол (7ГМР), выделенный из стандартизированных экстрактов хвойных деревьев (ель, пихта белая, сосна и др.), характеризуется хорошей биоусвояемостью и безопасностью. 7ГМР оказывает мультитаргетное воздействие на систему иммунитета, способствуя контролю воспаления через сигнальные каскады TNF-α/NF-κB (фактор некроза опухоли альфа/ядерный фактор каппа B; англ. tumor necrosis factor alpha/nuclear factor kappa B), модуляцию метаболизма лейкотриенов и простагландинов [1][2]. Онкопротекторные свойства 7ГМР активно изучаются [1]. В эксперименте 7ГМР (120 мг/кг/сут) ингибирует рост карциномы Эрлиха, причём даже на фоне стимулирования пролиферативных процессов эстрогенами [3]. Хемореактомный анализ 7ГМР позволил предположить, что онкопротекторные свойства молекулы связаны с ингибированием гемоксигеназы, циклинзависимых киназ и ряда ростовых факторов [2].
Современный подход к изучению фармакологического действия лекарственных средств предполагает постгеномную парадигму, в рамках которой исследуются не только эффекты молекул на ингибирование тех или иных белков протеома, но и воздействие молекул на транскриптом, т. е. на уровни мРНК, характеризующие степень экспрессии генов (рис. 1) [4]. Тогда как ингибирование белков протеома соответствует «тактическим эффектам» лекарственных средств (несколько часов), транскриптомные данные об эффектах воздействия лекарства позволяют оценивать долговременные, «стратегические» эффекты их использования, измеряющиеся днями и неделями [5]. Оценка более длительных эффектов воздействия лекарственных средств имеет особое значение для длительно применяющихся онкопротекторных препаратов.
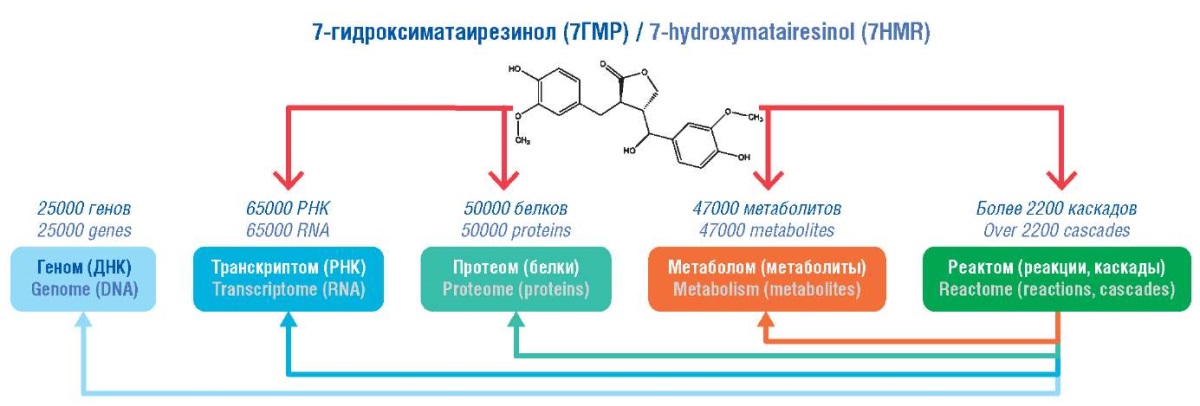
Рисунок 1. Фармакологическая активность 7-гидроксиматаирезинола
в постгеномной парадигме.
Figure 1. Pharmacological activity of 7-hydroxymatairesinol
in a post-genomic paradigm.
Цель: оценка транскриптомных эффектов лигнана 7ГМР по отношению к клеткам опухоли молочной железы.
Материалы и методы / Materials and Methods
Методика хемоинформационного исследования подробно описана в нашей предыдущей работе [6]. Так, для выбранного типа клеток (в случае настоящей работы – опухолевые клетки рака молочной железы, линия MCF7) и выбранного времени воздействия вещества на клетки (24 ч) в базе данных Gene Expression Omnibus (GEO; https://www.ncbi.nlm.nih.gov/geo/) находятся выборки данных транскриптомных исследований. На основании применения теорий и алгоритмов комбинаторного анализа хемографов [7–9] осуществляется моделирование воздействия молекул на экспрессию генов в изучаемой клеточной линии. Затем разработанные и верифицированные кросс-валидацией модели применяются к дозозависимому анализу эффектов исследуемой молекулы (т. е. 7ГМР). В результате формируются 2 списка генов: (1) гены, экспрессия которых повысилась в результате воздействия изучаемой молекулы по сравнению с контролем («Список+»), и (2) гены, экспрессия которых снизилась («Список–»). К этим двум спискам генов применяются алгоритмы биоинформационного анализа, в частности, метода функционального связывания [4] с использованием международной номенклатуры Gene Ontology (GO) по биологическим ролям генов/белков.
Методы статистического анализа / Statistical analysis
Сравнение частот различных биологических ролей генов в двух списках осуществлялось с использованием критериев параметрической и непараметрической статистики (χ², t-критерий Стьюдента) в пакете программ ChemStudyY2 (Россия) для хемотранскриптомного анализа, разработанных на Вычислительном центре РАН.
Результаты и обсуждение / Results and Discussion
Выделение групп генов с изменённой транскрипцией / Isolation of gene groups with altered transcription
Хемотранскриптомное изучение воздействия молекулы 7ГМР на клетки MCF7 (при 24-часовой экспозиции клеток) позволило выявить существенные изменения транскрипции (р < 0,05 по t-тесту; r = 0,50; величина изменений > 5 %/мкмоль 7ГМР) для 3468 из 12700 генов. При этом найдено снижение экспрессии («Список–») для 1387 генов и повышение экспрессии («Список+») для 2081 генов. Статистический анализ позволил выявить 56 GO-категорий функций 3468 генов, которые можно объединить в 6 групп, связанных с (1) деградацией белков на протеасомах, (2) процессами клеточной пролиферации, (3) рибосомальным биосинтезом белка, (4) поддержкой синтеза АТФ (энергетический метаболизм), (5) регуляцией вялотекущего системного воспаления и (6) собственно онкопротекторным действием (табл. 1, рис. 2). Очевидно, что экспрессия генов систематически снижается во всех группах, кроме группы «Онкопротекторное действие» (рис. 3), а частота встречаемости генов со сниженной экспрессией при воздействии 7ГМР («Список–») достоверно отличалась от частоты встречаемости генов с повышенной экспрессией при воздействии 7ГМР («Список+») (рис. 4).
Таблица 1. Категории международной номенклатуры Gene Ontology (GO).
Table 1. Categories of Gene Ontology (GO) international nomenclature.
|
Категория GO GO category |
Описание категории GO Category GO description |
n— |
n+ |
р |
|
Протеасомная деградация белков / Proteasomal protein degradation |
||||
|
[GO:0000502] |
протеасомный комплекс / proteasome complex |
34 |
1 |
2,1×10–8 |
|
[GO:0005839] |
ядро протеасомы / proteasome core |
12 |
1 |
0,002244 |
|
[GO:0019787] |
убиквитиновая трансфераза / ubiquitin transferase |
6 |
0 |
0,014233 |
|
[GO:0019773] |
альфа-субъединица протеасомы / proteasome alpha subunit |
5 |
0 |
0,025256 |
|
[GO:0045732] |
усиление катаболизма белков / increased protein catabolism |
13 |
4 |
0,028708 |
|
Пролиферация клеток / Сell proliferation |
||||
|
[GO:0032201] |
поддержание теломер, полуконсервативная репликация / maintenance of telomeres, semi-conservative replication |
34 |
0 |
4,7×10–9 |
|
[GO:0006297] |
ресинтез ДНК при ремонте ДНК / DNA resynthesis during DNA repair |
23 |
0 |
1,5×10–6 |
|
[GO:0006303] |
ремонт двухконцевых обрывов ДНК / repair of double-ended DNA breaks |
22 |
0 |
2,5×10–6 |
|
[GO:0006284] |
ремонт ДНК, вырезка поврежденных нуклеотидов / DNA repair, cutting out damaged nucleotides |
17 |
0 |
3,6×10–5 |
|
[GO:0003678] |
активность ДНК хеликазы / DNA helicase activity |
15 |
0 |
0,000104 |
|
[GO:0006362] |
транскрипция от промотора РНК-полимеразы I / transcription from the RNA polymerase I promoter |
14 |
0 |
0,000178 |
|
[GO:0006361] |
инициация транскрипции от промотеров РНКазы-1 / transcription initiation from RNase-1 promoters |
14 |
0 |
0,000178 |
|
[GO:0045739] |
позитивная регуляция репарации ДНК / positive regulation of DNA repair |
12 |
0 |
0,000522 |
|
[GO:0000082] |
G1/S переход при митозе / G1/S transition during mitosis |
59 |
8 |
3,3×10–10 |
|
[GO:0051301] |
деление клеток / cell division |
143 |
21 |
2,3×10–22 |
|
[GO:0000075] |
контрольная точка клеточного цикла / cell cycle checkpoint |
6 |
0 |
0,014233 |
|
[GO:0007064] |
когезия сестринских хроматид при митозе / cohesion of sister chromatids during mitosis |
6 |
0 |
0,014233 |
|
[GO:0035999] |
тетрагидрофолатная интерконверсия / tetrahydrofolate interconversion |
5 |
0 |
0,025256 |
|
[GO:0031497] |
сборка хроматина / chromatin assembly |
5 |
0 |
0,025256 |
|
[GO:0000723] |
поддержание теломер / maintenance of telomeres |
22 |
6 |
0,002411 |
|
Синтез белка / Рrotein synthesis |
||||
|
[GO:0070125] |
трансляция в митохондриях / translation in mitochondria |
40 |
2 |
3,8×10–9 |
|
[GO:0000375] |
сплайсинг мРНК / mRNA splicing |
14 |
0 |
0,000178 |
|
[GO:0005840] |
рибосома / ribosome |
12 |
0 |
0,000522 |
|
[GO:0005681] |
сплайсеосомный комплекс / spliceosome complex |
45 |
4 |
3,8×10–9 |
|
[GO:0030150] |
импорт белка в митохондриальный матрикс / protein import into the mitochondrial matrix |
8 |
0 |
0,004637 |
|
[GO:0006458] |
фолдинг белков / protein folding |
8 |
0 |
0,004637 |
|
[GO:0051131] |
шаперон-контролируемая сборка белковых комплексов / chaperone-controlled assembly of protein complexes |
8 |
0 |
0,004637 |
|
[GO:0003755] |
пептидилпролил-цис-транс-изомераза / peptidylprolyl-cis-trans-isomerase |
7 |
1 |
0,033717 |
|
[GO:0032543] |
митохондриальный синтез белка / mitochondrial protein synthesis |
11 |
3 |
0,032206 |
|
[GO:0006412] |
трансляция (синтез белка) / translation (protein synthesis) |
41 |
12 |
6,1×10–5 |
|
Энергетический метаболизм / Еnergy metabolism |
||||
|
[GO:0005747] |
митохондриальный комплекс I дыхательной цепи / mitochondrial complex I of the respiratory chain |
13 |
1 |
0,001315 |
|
[GO:0005750] |
митохондриальный комплекс III дыхательной цепи / mitochondrial complex III of the respiratory chain |
7 |
0 |
0,008095 |
|
[GO:0045334] |
клатриновый эндоцитотический везикул / clathrin endocytic vesicle |
7 |
0 |
0,008095 |
|
[GO:0006099] |
цикл трикарбоновой кислоты / tricarboxylic acid cycle |
6 |
0 |
0,014233 |
|
[GO:0032981] |
дыхательная цепь-1 митохондрий / mitochondria respiratory chain-1 |
16 |
3 |
0,002794 |
|
[GO:0006771] |
метаболизм рибофлавина / riboflavin metabolism |
5 |
0 |
0,025256 |
|
[GO:0033864] |
повышение активности НАДФ-оксидазы / increased activity of NADP-oxidase |
5 |
0 |
0,025256 |
|
[GO:0055089] |
гомеостаз жирных кислот / fatty acid homeostasis |
4 |
0 |
0,045392 |
|
[GO:0045254] |
комплекс пируватдегидрогеназы / pyruvate dehydrogenase complex |
4 |
0 |
0,045392 |
|
[GO:0055114] |
окислительно-восстановительный процесс / redox process |
24 |
10 |
0,015898 |
|
Хроническое воспаление / Сhronic inflammation |
||||
|
[GO:0033209] |
сигнальный путь TNF-α / TNF-α signaling pathway |
34 |
5 |
3,1×10–6 |
|
[GO:0051059] |
связывание NF-κB / NF-κB binding |
6 |
0 |
0,014233 |
|
[GO:0035722] |
интерлейкин-12-опосредованный сигнальный путь / interleukin-12-mediated signaling pathway |
18 |
5 |
0,006558 |
|
[GO:0043312] |
дегрануляция нейтрофилов / neutrophil degranulation |
90 |
59 |
0,009646 |
|
Онкопротекторное действие / Oncoprotective effect |
||||
|
[GO:0030593] |
хемотаксис нейтрофилов / neutrophil chemotaxis |
5 |
17 |
0,010304 |
|
[GO:0060337] |
сигнальный путь интерферона I / interferon I signaling pathway |
2 |
9 |
0,034559 |
|
[GO:0051898] |
отрицательная регуляция протеинкиназы В / downregulation of protein kinase B |
1 |
11 |
0,003839 |
|
[GO:0016525] |
отрицательная регуляция ангиогенеза / downregulation of angiogenesis |
1 |
17 |
0,000157 |
|
[GO:0006590] |
генерация тиреоидных гормонов / thyroid hormones generation |
0 |
5 |
0,025256 |
|
[GO:0043303] |
дегрануляция тучных клеток / mast cell degranulation |
0 |
4 |
0,045392 |
|
[GO:0042359] |
метаболизм витамина D / vitamin D metabolism |
0 |
5 |
0,025256 |
|
[GO:0002523] |
миграция лейкоцитов при воспалительной реакции / leukocytes migration during an inflammatory reaction |
0 |
6 |
0,014233 |
|
[GO:0001972] |
связывание ретиноидов / binding of retinoids |
0 |
4 |
0,045392 |
|
[GO:0048245] |
эозинофильный хемотаксис / eosinophilic chemotaxis |
0 |
4 |
0,045392 |
|
[GO:0046888] |
снижение секреции гормонов / decreased hormones secretion |
0 |
5 |
0,025256 |
|
[GO:0097327] |
отклик на цисплатин / response to cisplatin |
0 |
4 |
0,045392 |
Примечание: «n–» – число генов со сниженной экспрессией
при воздействии 7-гидроксиматаирезинола;
«n+» – число генов со повышенной экспрессией
при воздействии 7-гидроксиматаирезинола;
TNF-α – фактор некроза опухоли-альфа;
NF-κB – ядерный фактор каппа B;
р – статистическая значимость различий по критерию χ².
Note: "n–" – the number of genes with downregulated expression
upon exposure to 7-hydroxymatairesinol;
"n+" – the number of genes with upregulated expression
upon exposure to 7-hydroxymatairesinol;
TNF-α – tumor necrosis factor-alpha;
NF-κB – nuclear factor kappa B;
p – significance of differences according to the criterion χ².
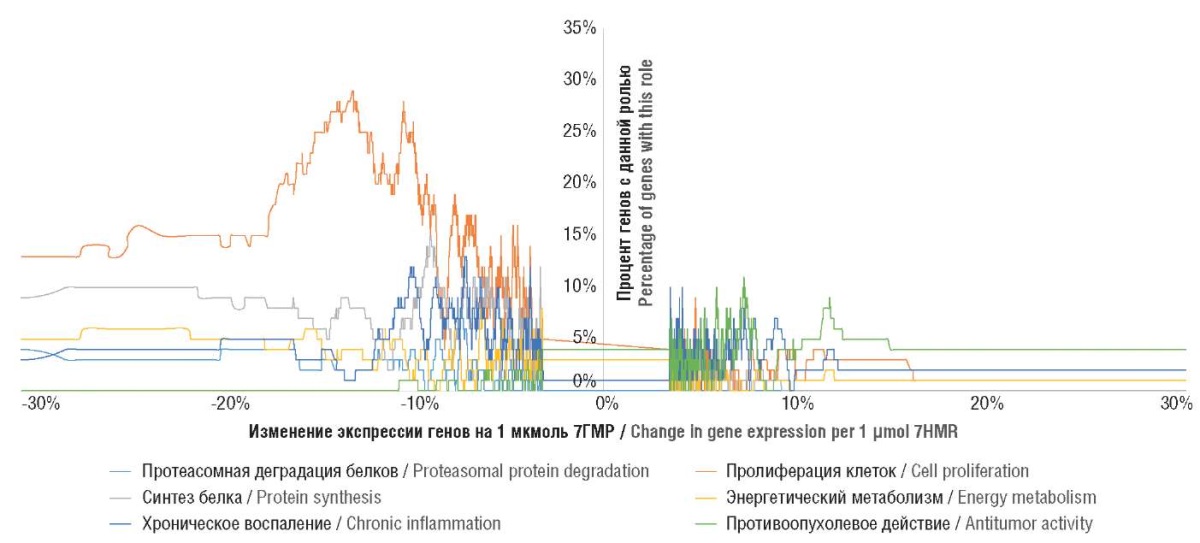
Рисунок 2. Профили частоты встречаемости
генов каждой из 6 функциональных групп,
экспрессия которых дозозависимо изменяется при воздействии
7-гидроксиматаирезинола (7ГМР) на клетки линии MCF7
(по результатам хемотранскриптомного анализа).
Figure 2. Dose-dependent change in frequency profiles for gene prevalence
in the 6 functional groups of 7-hydroxymatairesinol (7HMR)-exposed MCF7 cells
(based on chemotranscriptome analysis).
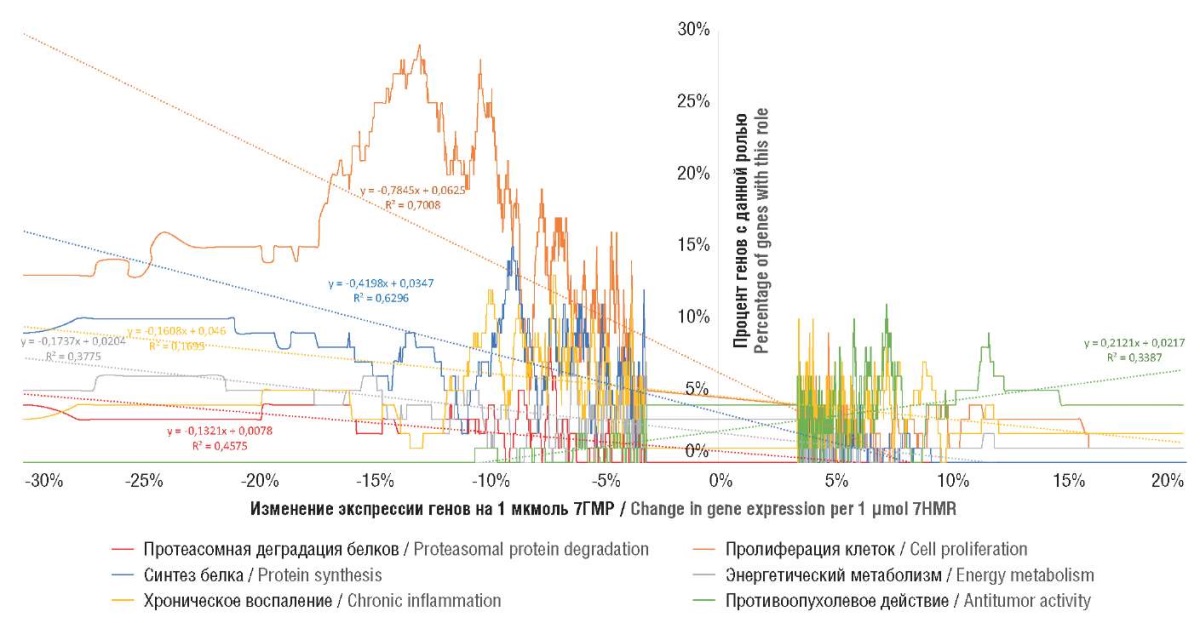
Рисунок 3. Линейные аппроксимации профилей частот встречаемости
генов каждой из 6 функциональных групп,
экспрессия которых дозозависимо изменяется при воздействии
7-гидроксиматаирезинола (7ГМР) на клетки линии MCF7
(по результатам хемотранскриптомного анализа).
Figure 3. Linear approximations of dose-dependent change
in frequency profiles for gene prevalence in the 6 functional groups
of 7-hydroxymatairesinol (7HMR)-exposed MCF7 cells
(based on chemotranscriptome analysis).
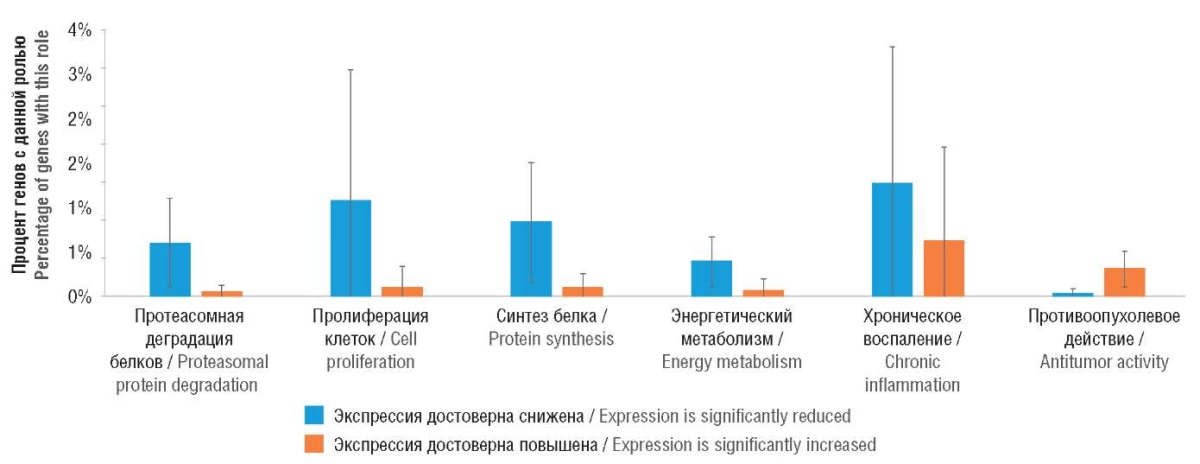
Рисунок 4. Попарное сравнение частот встречаемости
генов в 6 функциональных группах,
экспрессия которых дозозависимо изменяется при воздействии
7-гидроксиматаирезинола (7ГМР) на клетки линии MCF7
(по результатам хемотранскриптомного анализа).
Figure 4. Pairwise comparison of dose-dependent change
in frequency profiles for gene prevalence in the 6 functional groups
of 7-hydroxymatairesinol (7HMR)-exposed MCF7 cells
(based on chemotranscriptome analysis).
Сравнение групп генов с изменённой транскрипцией посредством GO-категорий / Comparison of transcriptionally altered gene groups using GO categories
На рисунке 5 представлены количества генов списков «Список–» и «Список+», соответствующие GO-категориям в таблице 1. В целом, гены с повышенной экспрессией вследствие воздействия 7ГМР значимо отличаются по своим биологическим ролям от генов со сниженной экспрессией. Совместное рассмотрение профилей частот (рис. 3) и данных на рисунке 5 указывает на несомненное повышение уровней транскриптов генов с онкопротекторным действием на фоне снижения экспрессии генов из 5 остальных функциональных групп.

Рисунок 5. Встречаемость функциональных групп генов со сниженной
и с повышенной экспрессией при 24-часовом воздействии
7-гидроксиматаирезинола (7ГМР) на клетки MCF7
(по результатам хемотранскриптомного анализа).
Примечание: n – количество соответствующих генов.
Figure 5. Prevalence of functional groups of genes with downregulated
and upregulated expression in MCF7 cells after 24-hour exposure
to 7-hydroxymatairesinol (7HMR)
(according to chemotranscriptome analysis).
Note: n – number of corresponding genes.
Молекулярно-физиологическая интерпретация результатов / Molecular and physiological interpretation of the results
Молекулярно-физиологическая интерпретация данных хемотранскриптомного анализа молекулы 7ГМР основана на 6 основных группах результатов:
A. снижение интенсивности внутриклеточного гомеостаза белков;
B. снижение экспрессии генов, вовлеченных в энергетический метаболизм;
C. снижение экспрессии генов, вовлечённых в онкопролиферацию;
D. снижение экспрессии генов, вовлечённых в хроническое воспаление;
E. повышение экспрессии онкопротекторных генов;
F. повышение экспрессии аскорбат-зависимых генов.
А. Снижение транскрипции генов синтеза белка (194 гена) и протеолиза белка (70 генов) указывает на общее снижение интенсивности белкового обмена под воздействием 7ГМР. Снижение синтеза белка, включая процессы сплайсинга мРНК и транспорта белков, указывает на уменьшение количеств ростовых факторов. Снижение протеолиза белков соответствует снижению хронического воспаления, регулируемого через сигнальный путь NF-κB, и активации апоптоза.
B. Снижение транскрипции генов энергетического метаболизма, включая белковые комплексы дыхательной цепи, цикла Кребса, пируватдегидрогеназы и других, указывает на уменьшение обеспеченности онкопролиферативных процессов молекулами АТФ посредством 7ГМР.
C. Снижение транскрипции онкопролиферативных генов (теломеры, репликации ДНК, клеточного цикла и прочих) соответствует уменьшению числа возможных делений опухолевых клеток и преодолению резистентности к химиотерапии [6] при воздействии 7ГМР.
D. Снижение транскрипции генов хронического воспаления способствует поддержке противовоспалительных эффектов 7ГМР на уровне протеома человека [1], в частности, ингибирование NF-κB за счёт торможения протеолиза киназы Iκ-B.
E. Повышение под воздействием 7ГМР транскрипции более 100 онкопротекторных генов затрагивает столь разнообразные процессы, как:
- снижение хемотаксиса нейтрофилов, способствующих прогрессии опухолей молочной железы [14], и повышение хемотаксиса Т-лимфоцитов, поддерживающих естественную онкопротекторную реакцию организма [15];
- активность сигнального пути интерферона, который стимулирует апоптоз опухолевых клеток [16];
- отрицательную регуляцию протеинкиназы В, что ингибирует рост опухолевых клеток MCF7 [17];
- отрицательную регуляцию ангиогенеза, что способствует замедлению роста опухолей;
- синтез тиреоидных гормонов, которые ингибируют рост и выживаемость опухолевых клеток, в том числе клеток линии MCF7 [18];
- хемотаксис эозинофилов, инфильтрация и дегрануляция которых внутри опухолей способствует разрушению последних [19];
- поддержку метаболизма витамина D, который является одним из важнейших онкопротекторных микронутриентов [20];
- повышение экспрессии рецептора ретиноидов RXR-α, регулирующего пролиферацию, дифференциацию и апоптоз клеток (активация рецептора RXR-αвызывает апоптоз опухолевых клеток MCF7 [21]);
- повышение экспрессии гена ABCC2, кодирующего канал-транспортер цисплатина и др.
F. Повышение транскрипции аскорбат-зависимых генов является важным аспектом воздействия 7ГМР, так как аскорбиновая кислота проявляет выраженные онкопротекторные свойства [22][23]. Анализ встречаемости белковых кофакторов (табл. 2) показал, что 7ГМР способствовал преимущественному снижению экспрессии генов, которые кодируют белки с такими кофакторами, как железо-серные кластеры [ 4Fe–4S] (15 генов; р = 0,0015), ионы магния (80 генов; р = 0,001), калий (6 генов; р = 0,014) и железо (4 гена; р = 0,045). Все перечисленные кофакторы играют важную роль в поддержании «клеточного дыхания» и энергетического метаболизма опухолевых клеток, так что снижение их экспрессии соответствует онкопротекторному действию. Кроме того, 7ГМР повышал транскрипцию генов, белки которых содержат аскорбат-анион в качестве кофактора (6 генов; р = 0,058). Частота встречаемости генов со сниженной экспрессией при воздействии 7ГМР («Список–») достоверно отличалась от частоты встречаемости генов с повышенной экспрессией при воздействии 7ГМР («Список+»).
Таблица 2. Кофакторы белков.
Table 2. Protein cofactors.
|
Кофактор / Cofactor |
n— |
n+ |
р |
|
[ 4Fe–4S] |
15 |
2 |
0,001579 |
|
Mg2+ |
80 |
48 |
0,004043 |
|
K+ |
6 |
0 |
0,014233 |
|
Fe2+ |
4 |
0 |
0,045392 |
|
Аскорбат-анион / Ascorbate anion |
1 |
6 |
0,058561 |
Примечание: «n–» – число генов со сниженной экспрессией
при воздействии 7-гидроксиматаирезинола;
«n+» – число генов со повышенной экспрессией
при воздействии 7-гидроксиматаирезинола;
р – статистическая значимость различий по критерию χ².
Note: "n–" – the number of genes with downregulated expression
upon exposure to 7-hydroxymatairesinol;
"n+" – the number of genes with upregulated expression
upon exposure to 7-hydroxymatairesinol;
p – significance of differences according to the criterion χ².
Примеры дозозависимого изменения экспрессии генов под воздействием 7ГМР / Examples
of dose-dependent changes in gene expression upon exposure to 7HMR
В таблице 3 приведены отдельные примеры дозозависимого изменения экспрессии генов под воздействием 7ГМР, полученные в результате проведения хемотранскриптомного анализа молекулы.
Таблица 3. Примеры дозозависимого изменения экспрессии генов
под воздействием 7-гидроксиматаирезинола
по результатам хемотранскриптомного анализа.
Table 3. Examples of dose-dependent changes in gene expression
upon exposure to 7-hydroxymatairesinol
according to the results of chemotranscriptomal analysis.
|
Ген / Gene |
ИЭ, % / ЕС, % |
Белок / Protein |
Функция белка / Protein function |
|
CDCA3 |
–18,5 |
Белок А3 цикла деления клеток / Replication protein A3 |
Инициация деления клеток, фаза G2/M клеточного цикла / Initiation of cell division, G2/M phase of the cell cycle |
|
UBE3C |
–30,4 |
Убиквитин протеин лигаза E3C / Ubiquitin protein ligase E3C |
Деградация белков на протеасоме / Protein degradation on the proteasome |
|
RUVBL1 |
–16,3 |
RuvB-подобный белок-1 / RuvB-like protein-1 |
Активация онкогенов и протоонкогенов, репарация ДНК / Activation of oncogenes and proto-oncogenes, DNA repair |
|
PSMB3 |
–15,9 |
Субъединица протеасомы бета-3 / Proteasome subunit beta-3 |
Компонент протеасомы, участвует в протеолизе внутриклеточных белков / Proteasome component involved in the proteolysis of intracellular proteins |
|
DCLRE1B |
–7,6 |
Белок-1B ремонта ДНК / Protein-1B DNA repair |
Защита теломер во время S-фазы клеточного цикла / Telomere protection during the S-phase of the cell cycle |
|
DKC1 |
–6,7 |
Дискерин / Diskerin |
Поддержание теломер / Maintenance of telomeres |
|
NHP2 |
–6,3 |
Рибонуклеопротеин NHP2 / Ribonucleoprotein NHP2 |
Поддержание теломер / Maintenance of telomeres |
|
DDIT4 |
16,2 |
Транскрипт 4, индуцируемый повреждением ДНК / Transcript 4 induced by DNA damage |
Регулирует пролиферацию посредством ингибирования рапамицинового комплекса 1 (белковый комплекс mTORC1) / Regulates proliferation through inhibition of rapamycin complex 1 (mTORC1 protein complex) |
|
IFNAR2 |
14,7 |
Интерферон рецептор-2 / Interferon receptor subunit-2 |
Рецептор-1 интерферонов альфа и бета / Interferon alpha and beta receptor-1 |
|
SLURP1 |
13,1 |
Белок SLURP1 с доменом LY6/PLAUR / SLURP1 protein with LY6/PLAUR domain |
Участвует в поддержании физиологической и структурной целостности слоев кератиноцитов / Involved in maintaining the physiological and structural integrity of keratinocyte layers |
|
SZT2 |
12,6 |
Белок SZT2 (судорожный порог-2) / SZT2 protein (seizure threshold-2) |
Ингибирует активность mTORC1, клеточный ответ на окислительный стресс / Inhibits mTORC1 activity, cellular response to oxidative stress |
|
ITIH5 |
10,6 |
Ингибитор интер-α-глобулина H5 / Inter-α-globulin H5 inhibitor |
Подавляет развитие опухолей / Suppresses of tumors development |
Примечание: ИЭ – изменение экспрессии на 1 мкмоль 7-гидроксиматаирезинола.
Note: EС – expression change per 1 µmol of 7-hydroxymatairesinol.
Как было отмечено выше, 7ГМР может приводить к снижению экспрессии генов, вовлечённых в протеасомную деградацию белков (протеолиз). Например, снижалась экспрессия гена PSMB3, кодирующего субъединицу бета-3 протеасомы, и также гена UBE3C, кодирующего убиквитин протеин лигазу E3C, которая направляет белки на деградацию на протеасоме [24]. Снижение экспрессии гена CDCA3 (белок А3 цикла деления клеток) соответствует торможению деления клеток (митоза), так как данный белок опосредует убиквитинирование и деградацию WEE1-киназы в фазе G2/M клеточного цикла, тем самым инициируя митоз.
Хемотранскриптомный анализ показал, что 7ГМР снижает экспрессию генов, белки которых участвуют в пролиферации опухолевых клеток. Например, снижается экспрессия гена RUVBL1, который экcпреccируется во всех видах клеток. Ген RUVBL1 кодирует ДНК-геликазу – фермент под названием «RuvB-подобный белок-1», разъединяющий нити двойной спирали ДНК, которая участвует в транскрипционной активации генов. Белок RUVBL1 необходим для активации транскрипционных программ онкогенов и протоонкогенов, стимулирующих рост опухоли, а также для репарации ДНК. RuvB-подобный белок-1 играет существенную роль в онкогенной трансформации, вызываемой геном MYC, который принципиально необходим для пролиферации опухолевых клеток [25].
7ГМР может снижать экспрессию генов, белки которых участвуют в поддержании структуры теломер – концевых участков хромосом, которые укорачиваются в каждом цикле деления клетки, что является одной из важнейших причин биологического старения. Поддержание структуры теломер осуществляется теломеразой – ферментным комплексом, добавляющим специфическую последовательность ДНК к теломерам, тем самым стабилизируя хромосомы [26].
В частности, хемореактомный анализ 7ГМР указал на снижение экспрессии гена DKC1 (дискерин, необходим для биогенеза рибосома и поддержания теломер, пролиферации клеток [27]) и гена DCLRE1B (белок-1B перекрестного ремонта ДНК, осуществляющего поддержание и защиту теломер во время S-фазы клеточного цикла). Белок, кодируемый геном DCLRE1B, необходим для формирования теломерной петли (Т-петли), защищает теломеры от ферментов ремонта ДНК, от повреждения во время репликации при участии топоизомераз TOP1, TOP2A и TOP2B [28].
Под воздействием 7ГМР может снижаться экспрессия гена NHP2. Соответствующий этому гену рибонуклеопротеин NHP2 требуется для поддержания теломер, участвуя в модификациях и внутриядерном транспорте белка TERC – РНК-компонента обратной транскриптазы ферментного комплекса теломеразы [29]. Избыточная активация теломеразы наблюдается в 90 % всех раковых заболеваний человека [30], что указывает на важность этого механизма в формировании и развитии опухолевых заболеваний.
Хемореактомный анализ указал на повышение под воздействием 7ГМР экспрессии генов, подавляющих развитие опухолей: гена ITIH5 (ингибитор интер-α-глобулина H5, экспрессируется в молочных железах и яичниках, подавляет развитие опухолей), гена SLURP1 (белок SLURP1, поддерживает целостность слоев кератиноцитов [31], маркер поздней дифференциации кожи, обладает онкопротекторной активностью [32]), гена IFNAR2 (рецептор интерферонов альфа и бета, участвует в IFN-опосредованной активации сигнальных белков STAT1, STAT2 и STAT3 [33]) и гена SZT2 (белок SZT2, ингибирует активность mTORC1, участвует в регуляции mTORC1 глюкозой [34], играет роль в клеточном ответе на окислительный стресс [35]).
Полученные результаты указывают на дальнейшие перспективы исследований молекулы 7ГМР. Во-первых, необходимо установить, являются ли описанные изменения в транскрипции генов результатом воздействия 7ГМР на некоторые факторы транскрипции, которые одновременно являются таргетными белками для 7ГМР. Во-вторых, следует установить, являются ли описанные выше изменения транскрипции результатом изменения сигнального состояния клеток под воздействием 7ГМР.
Результаты хемотранскриптомного анализа указывают на реалистичные механизмы онкопротекторного действия 7ГМР. Состояние гиперэстрогении (например, при мастопатии) соответствует гиперпролиферативному профилю пациентки. Обычно считается, что 7ГМР якобы является «прямым антагонистом» эстрогенов. Однако прямых взаимодействий 7ГМР с рецепторами эстрогенов не было установлено. В то же время хемотранскриптомный анализ указал на целый комплекс физиологических свойств 7ГМР, которыми данная молекула противодействует проопухолевым свойствам эстрогенов (регуляция экспрессии более 100 генов онкопротекторного действия, 401 гена пролиферации клеток, 148 генов хронического воспаления и др.).
В экспериментальных и клинических исследованиях были продемонстрированы онкопротекторные свойства 7ГМР в условиях гиперэстрогении. Приём 120 мг/сут 7ГМР достоверно тормозил интенсивность роста опухолевых узлов на фоне гиперэстрогении у мышей с моделью карциномы Эрлиха [3]. В группе пациенток с фиброзно-кистозной мастопатией в постменопаузальном периоде (n = 60) были получены количественные оценки влияния приёма 7ГМР в режиме 60 мг/сут, 1 мес на показатели эстрогенового баланса. Приём 7ГМР способствовал нормализации метаболизма эстрогенов (снижение уровней эстрадиола, эстрона, эстриола, 16а-ОНЕ1) и улучшению клинической симптоматики мастопатии, тем самым снижая опухолевые риски, связанные с гиперэстрогенией [3].
Заключение / Conclusion
В настоящей работе представлены результаты хемотранскриптомного исследования воздействий 7ГМР на транскрипцию более 12 тыс. генов человека в линии опухолевых клеток MCF7 (рак молочной железы). Полученные результаты позволили не только описать сложную систему взаимодействий 7ГМР с транскриптомом, но и сформулировать новые механизмы онкопротекторного действия 7ГМР.
Результаты хемотранскриптомного анализа молекулы 7ГМР при оценке влияния на опухолевые клетки линии MCF7 указали на несколько важных особенностей ее транскриптомного воздействия. В частности, под воздействием 7ГМР может снижаться экспрессия онкопролиферативных генов (n = 401), генов синтеза белка (n = 194) и протеолиза белка (n = 70), синтеза АТФ в опухолевых клетках (n = 91) и регуляции хронического воспаления (n = 148). Уменьшение экспрессии этих групп генов тормозит процессы пролиферации и жизнедеятельности опухолевых клеток, одновременно защищая организм от избыточного воспаления. Параллельно 7ГМР способствовал преимущественному повышению транкрипции групп генов, вовлеченных в онкопротекторное действие (более 100 генов), в том числе генов, участвующих в поддержании онкопротекторного иммунитета, а также генов, опосредующих онкопротекторные свойства витамина D, ретиноидов и витамина С. Описанные изменения в транскрипции генов усиливают свойства воздействия лигнанов на белки протеома и указывают на перспективы использования 7ГМР у пациенток с опухолевыми заболеваниями.
Хемотранскриптомный анализ эффектов молекулы 7-гидроксиматаирезинола на опухолевые клетки молочной железы человека линии MCF7
Список литературы
1. Громова О.А., Торшин И.Ю., Рубашкина А.Н. и др. Систематический анализ фундаментальных и клинических исследований лигнана 7-гидроксиматаирезинола. Эффективная фармакотерапия. 2019;15(13):34–41. https://doi.org/10.33978/2307-3586-201915-13-34-41.
2. Торшин И.Ю., Рубашкина А.Н., Лапочкина Н.П., Громова О.А. Хемореактомный анализ 7-гидроксиматаирезинола, 17-эстрадиола, фитоэстрогена β-ситостирола и эпигаллокатехин-3галлата. Акушерство, Гинекология и Репродукция. 2020;14(3):347–60. https://doi.org/10.17749/2313-7347/ob.gyn.rep.2020.152.
3. Рубашкина А.Н., Лапочкина Н.П., Торшин И.Ю., Громова О.А. Роль 7гидроксиматаирезинола в модуляции метаболизма эстрогенов и терапии мастопатии. Гинекология. 2020;22(4):43–8. https://doi.org/10.26442/20795696.2020.4.200183.
4. Torshin I.Yu. Sensing the change from molecular genetics to personalized medicine. In: "Bioinformatics in the Post-Genomic Era" series. NY, USA: Nova Biomedical Books, 2009. 366 p. ISBN 1-60692-217-0.
5. Торшин И.Ю., Громова О.А. Экспертный анализ данных в молекулярной фармакологии. М., 2012. 684 с.
6. Торшин И.Ю., Громова О.А., Фролова Д.Е. и др. Дозозависимый хемотранскриптомный анализ дифференциального действия витамина D на экспрессию генов в клетках-предшественниках нейронов NPC и в опухолевых клетках MCF7 человека. Фармакокинетика и фармакодинамика. 2018;(2):35–51. https://doi.org/10.24411/2587-78362018-10013.
7. Torshin I. Yu., Rudakov K.V. On the application of the combinatorial theory of solvability to the analysis of chemographs. Part 1: fundamentals of modern chemical bonding theory and the concept of the chemograph. Pattern Recognition and Image Analysis. Advances in Mathematical Theory and Applications. 2014;24(1):11–23. https://doi.org/10.1134/S1054661814010209.
8. Торшин И.Ю., Громова О.А., Федотова Л.Э. и др. Хемоинформационный анализ молекулы оротовой кислоты указывает на противовоспалительные, нейропротекторные и кардиопротекторные эффекта лиганда магния. Фарматека. 2013;(13): 95–104.
9. Громова О.А., Торшин И.Ю., Федотова Л.Э., Громов А.Н. Хемореактомный анализ сукцината этилметилгидроксипиридина. Неврология, нейропсихиатрия, психосоматика. 2016;8(3):53–60. https://doi.org/10.14412/2074-2711-2016-3-53-60.
10. Громова О.А., Торшин И.Ю., Федотова Л.Э. Геронтоинформационный анализ эффект молекулы мексидола. Неврология, нейропсихиатрия, психосоматика. 2017;9(4):46–54. https://doi.org/10.14412/2074-2711-2017-4-46-54.
11. Torshin I.Y. The study of the solvability of the genome annotation problem on sets of elementary motifs. Pattern Recognition and Image Analysis. Advances in Mathematical Theory and Applications. 2011;21(4):652–62. https://doi.org/10.1134/S1054661811040171.
12. Torshin I.Y., Rudakov K.V. Combinatorial analysis of the solvability properties of the problems of recognition and completeness of algorithmic models. Part 1: factorization approach. Pattern Recognition and Image Analysis. Advances in Mathematical Theory and Applications. 2017;27(1):16–28. https://doi.org/10.1134/S1054661817010151.
13. Torshin I.Y., Rudakov K.V. On the application of the combinatorial theory of solvability to the analysis of chemographs. Part 2: local completeness of invariants of chemographs in view of the combinatorial theory of solvability. Pattern Recognition and Image Analysis. Advances in Mathematical Theory and Applications. 2014;24(2):196–208. https://doi.org/10.1134/S1054661814020151.
14. Mouchemore K.A., Anderson R.L., Hamilton J.A. Neutrophils, G-CSF and their contribution to breast cancer metastasis. FEBS J. 2018;285(4):665–79. https://doi.org/10.1111/febs.14206.
15. Mohammed Z.M., Going J.J., Edwards J. et al. The relationship between components of tumour inflammatory cell infiltrate and clinicopathological factors and survival in patients with primary operable invasive ductal breast cancer. Br J Cancer. 2012;107(5):864–73. https://doi.org/10.1038/bjc.2012.347.
16. Wang Y., Yu X., Song H. et al. The STAT-ROS cycle extends IFN-induced cancer cell apoptosis. Int J Oncol. 2018;52(1):305–13. https://doi.org/10.3892/ijo.2017.4196.
17. Li J., Zhang J., Jin L. et al. Silencing lnc-ASAH2B-2 inhibits breast cancer cell growth via the mTOR pathway. Anticancer Res. 2018;38(6):3427–34. https://doi.org/10.21873/anticanres.
18. Rogowski M., Gollahon L., Chellini G., Assadi-Porter F.M. Uptake of 3-iodothyronamine hormone analogs inhibits the growth and viability of cancer cells. FEBS Open Bio. 2017;7(4):587– 601. https://doi.org/10.1002/2211-5463.12205.
19. Furbert-Harris P.M., Laniyan I., Harris D. et al. Activated eosinophils infiltrate MCF-7 breast multicellular tumor spheroids. Anticancer Res. 2003;23(1A):71–8.
20. Громова О.А., Торшин И.Ю. Витамин D. Смена парадигмы. Под ред. Е.И. Гусева, И.Н. Захаровой. М.: ГЭОТАР-Медиа, 2017. 568 с.
21. Aouad P., Saikali M., Abdel-Samad R. et al. Antitumor activities of the synthetic retinoid ST1926 in two-dimensional and three-dimensional human breast cancer models. Anticancer Drugs. 2017;28(7):757–70. https://doi.org/10.1097/CAD.0000000000000511.
22. Громова О.А., Торшин И.Ю., Филимонова М.В., Сорокина М.А. Роль витаминов в профилактике рака и их влияние на эффективность онкопротекторной терапии: систематический анализ доказательных исследований. Терапия. 2018;(4):108–20.
23. Громова О.А., Торшин И.Ю., Мартынов А.И. и др. Систематический анализ применения витаминов в рамках многопрофильного стационара. Терапия. 2017;(6):89–99.
24. Wang M., Cheng D., Peng J., Pickart C.M. Molecular determinants of polyubiquitin linkage selection by an HECT ubiquitin ligase. EMBO J. 2006;25(8):1710–19. https://doi.org/10.1038/sj.emboj.7601061.
25. Grieb B.C., Gramling M.W., Arrate M.P. et al. Oncogenic protein MTBP interacts with MYC to promote tumorigenesis. Cancer Res. 2014;74(13):3591–602. https://doi.org/10.1158/00085472.CAN-13-2149.
26. Olovnikov A.M. A theory of marginotomy. The incomplete copying of template margin in enzymic synthesis of polynucleotides and biological significance of the phenomenon. J Theor Biol. 1973;41(1):181–90. https://doi.org/10.1016/0022-5193(73)90198-7.
27. Heiss N.S., Bachner D., Salowsky R. et al. Gene structure and expression of the mouse dyskeratosis congenita gene, dkc1. Genomics. 2000;67(2):153–63. https://doi.org/10.1006/geno.2000.6227.
28. Ye J., Lenain C., Bauwens S. et al. TRF2 and apollo cooperate with topoisomerase 2alpha to protect human telomeres from replicative damage. Cell. 2010;142(2):230–42. https://doi.org/10.1016/j.cell.2010.05.032.
29. Wang C., Meier U.T. Architecture and assembly of mammalian H/ACA small nucleolar and telomerase ribonucleoproteins. EMBO J. 2004;23(8):1857–67. https://doi.org/10.1038/sj.emboj.7600181.
30. Jafri M.A., Ansari S.A., Alqahtani M.H., Shay J.W. Roles of telomeres and telomerase in cancer, and advances in telomerase-targeted therapies. Genome Med. 2016;8(1):69. https://doi.org/10.1186/s13073-016-0324-x.
31. Favre B., Plantard L., Aeschbach L. et al. SLURP1 is a late marker of epidermal differentiation and is absent in Mal de Meleda. J Invest Dermatol. 2007;127(2):301–8. https://doi.org/10.1038/sj.jid.5700551.
32. Ridge R.J., Sloane N.H. Partial N-terminal amino acid sequence of the anti-neoplastic urinary protein (ANUP) and the anti-tumour effect of the N-terminal nonapeptide of the unique cytokine present in human granulocytes. Cytokine. 1996;8(1):1–5. https://doi.org/10.1006/cyto.1996.0001.
33. Duncan C.J., Mohamad S.M., Young D.F. et al Human IFNAR2 deficiency: Lessons for antiviral immunity. Sci Transl Med. 2015;7(307):307ra154. https://doi.org/10.1126/scitranslmed.aac4227.
34. Wolfson R.L., Chantranupong L., Wyant G.A. et al. KICSTOR recruits GATOR1 to the lysosome and is necessary for nutrients to regulate mTORC1. Nature. 2017;543(7645):438–42. https://doi.org/10.1038/nature21423.
35. Peng M., Yin N., Li M.O. SZT2 dictates GATOR control of mTORC1 signalling. Nature. 2017;543(7645):433–7. https://doi.org/10.1038/nature21378.
Об авторах
И. Ю. ТоршинРоссия
Торшин Иван Юрьевич – к.ф-м.н., к.х.н., старший научный сотрудник
Scopus Author ID: 7003300274
Author ID: 54104
WOS ID: C-7683-2018
119333 Москва, ул. Вавилова, д. 44
А. Н. Рубашкина
Россия
Рубашкина Анна Николаевна – аспирант кафедры онкологии, акушерства и гинекологии
153012 Иваново, Шереметевский проспект, д. 8
О. А. Громова
Россия
Громова Ольга Алексеевна – д.м.н., профессор, ведущий научный сотрудник
119333 Москва, ул. Вавилова, д. 44
Рецензия
Для цитирования:
Торшин И.Ю., Рубашкина А.Н., Громова О.А. Хемотранскриптомный анализ эффектов молекулы 7-гидроксиматаирезинола на опухолевые клетки молочной железы человека линии MCF7. Акушерство, Гинекология и Репродукция. 2023;17(5):584-596. https://doi.org/10.17749/2313-7347/ob.gyn.rep.2023.409
For citation:
Torshin I.Yu., Rubashkina A.N., Gromova O.A. Chemotranscriptomic analysis of 7-hydroxymatairesinol-related effects on MCF7 human breast tumor cells. Obstetrics, Gynecology and Reproduction. 2023;17(5):584-596. (In Russ.) https://doi.org/10.17749/2313-7347/ob.gyn.rep.2023.409

Контент доступен под лицензией Creative Commons Attribution-NonCommercial-ShareAlike 4.0 International License.











































